Windows10でNGS解析をやってみる - Bash on Ubuntu on Windows × Bioconda
概要
次世代シーケンス(NGS)のデータ解析というとLinux OS上で作業するのが一般的で、Windowsで行うためにはVMwareやVirtualBoxでLinuxの仮想環境を構築する必要がありました。
ただ、この方法だと環境構築が面倒で、場合によってはうまく動かないケースがありました。また、LinuxとWindowsの両方のシステムを動かすことになるので、パソコンへの負荷が大きいのも欠点でした。
しかし、2016年8月3日に配信開始された大型アップデート「Windows 10 Anniversary Update」で状況が変わりました。
このアップデートでLinux(Ubuntu)がWindows上で使用可能になる「Windows Subsystem for Linux (WSL)」が追加され、「Bash on Ubuntu on Windows」と呼ばれるBashがWindows上でも使えるようになりました。
今回はこのBash on Ubuntu on WindowsとBiocondaを組み合わせることで、NGSのデータ解析を行ってみたいと思います。
内容
1. Bash on Ubuntu on Windowsのインストール
下記のブログ記事ですでに紹介されているのでそちらを参照のこと。
インストール後、以下のようにメニュー画面からBash on Ubuntu on Windowsを呼び出すことができるようになります。

起動するとこんな感じです。

2. Biocondaのインストール
以前のブログで記事ですでに紹介したのでそちらを参照のこと。
Bash on Ubuntu on Windows上でもminicondaやBiocondaをインストールすることができます。

Bash on Ubuntu on WindowsからWindows内のフォルダにアクセスする
Bash on Ubuntu on Windows上からだと、WindowsのCドライブなどが/mnt上にマウントされているように見えます。

$ cd /mnt/c $ cd /mnt/d
などとそれぞれのドライブにアクセスできます。
もちろん、Windowsのシステムファイルに直接アクセスすることはできませんが、ファイル操作などは行うことができます。 そのため、LinuxのサブシステムとWindows10の間でのファイル共有も容易です。
$ touch test.txt $ mv test.txt /mnt/d

RNA-seqのデータをいじってみる
Biocondaを使って、sra-toolsとfastqcをインストールします。
$ conda install sra-tools $ conda install fastqc
今回、下記のRNA-seqのSRAファイルを使用する。
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSM1606099
Bashのホームディレクトリではなく、Dドライブ上にデータを配置するため、Dドライブに移動する。
cd /mnt/d
Bash on Ubuntu on Windows上でSRAファイルをダウンロードする。
$ wget ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByExp/sra/SRX/SRX870/SRX870796/SRR1795409/SRR1795409.sra
SRAファイルを解凍してFASTQファイルを用意する。
fastq-dump SRR1795409.sra
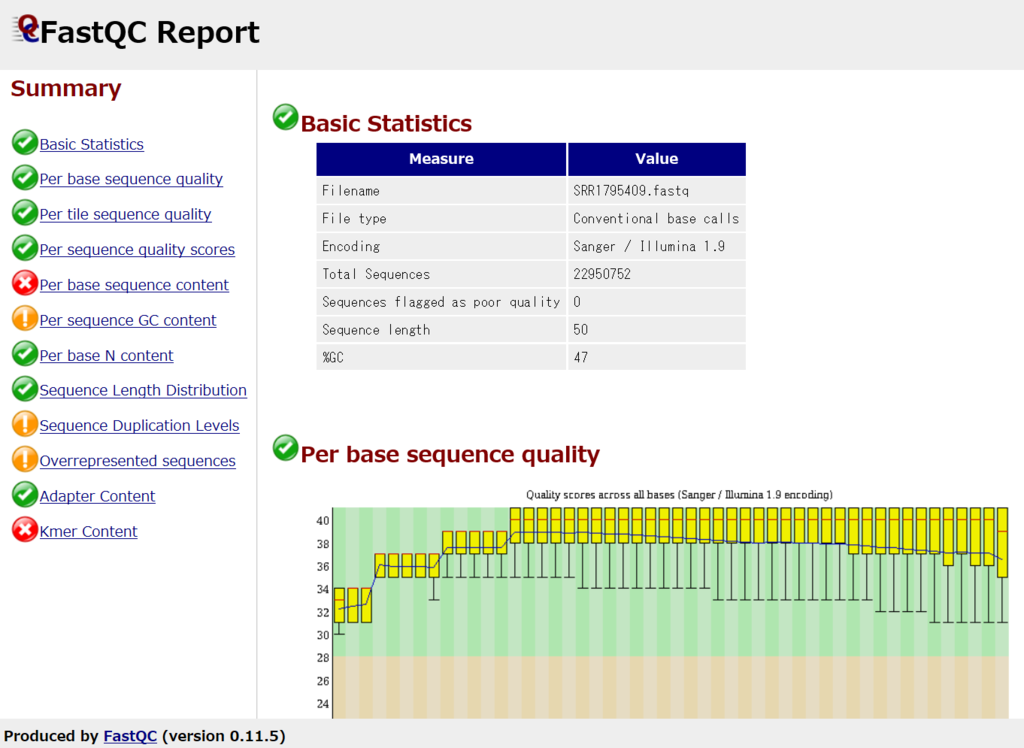
FastQCを使って、FASTQファイルの中身をチェックする。
fastqc SRR1795409.fastq
Dドライブ上にFastQCのデータが保存されているので、Windows上からアクセスしてファイルの中身を見ることができます。

こんな感じ。
ガッツリ解析しようと思ったらスパコンを利用するほうが良いと思いますが、Windows上にテスト環境を作って練習するには手軽かもしれません。