Pythonパッケージ: 便利なコマンドラインパーサClickを使ってみる
概要
コマンドラインパーサとして、argparseと呼ばれるPythonの標準パッケージが存在するが、使い勝手がいまいちだと感じています(あくまで個人的な感想ですが…)。
今回は、たまたま見つけたclickと呼ばれるサードパーティ製のPythonパッケージを用いてコマンドラインの引数を解釈するコードを書いてみようと思います。
argparseと比較してclickの書き方だと、コードが書きやすく、あとから読み返しやすくなると感じます。
Clickのドキュメントはこちら。
http://click.pocoo.org/6/
内容
すでに、こちらのブログに詳しく解説されているのでここでは忘備録的な記載に留めようと思います。
インストール
AnacondaもしくはBiocondaがインストールされていることが前提。
conda install click
基本的な使い方
@click.command()デコレータを用いて関数を修飾するだけでOK。
print関数の代わりにclick.echoを用いて文字列を標準出力することもできる。
@click.command()
def cmd():
click.echo("Command was Done.")
続けて、@click.option()デコレータに任意のオプションを追加していくことができる。argparseと同様に、-iや--ifastqなどのような-から始まるコマンドライン引数を指定することができる。
@click.command()
@click.option('-i', '--ifastq')
def cmd():
click.echo("Command was Done.")
また、作成したオプションに代入された値を取得するために、ifastqのように-を付けない形で文字列を指定することで、関数の中で値が代入された変数として扱えます。
以下のように、cmd関数の引数にifastqを指定しておく必要がある。
@click.command()
@click.option('-i', '--ifastq', 'ifastq')
def cmd(ifastq):
click.echo(ifastq)
helpでHelpが呼び出されたときの説明文を指定できる。
@click.command()
@click.option('-i', '--ifastq', 'ifastq', help='Input file in FASTQ file.')
def cmd(ifastq):
click.echo(ifastq)
実行例
#/usr/bin/env python
import click
@click.command()
@click.option('-i', '--ifastq', 'ifastq', help='Input file in FASTQ file.')
@click.option('-o', '--output-prefix', 'output', help='Prefix of output file.')
def cmd(ifastq, output):
click.echo(ifastq)
def main():
cmd()
if __name__ == '__main__':
main()
実行すると、
$ python test.py -i test.fastq test.fastq $ python test.py --help Usage: test.py [OPTIONS] Options: -i, --ifastq TEXT Input file in FASTQ file. -o, --output-prefix TEXT Prefix of output file. --help Show this message and exit.
コマンドライン引数のタイプを指定したい
INT型やFLOAT型など、引数のタイプがあらかじめわかっている場合、引数のタイプを指定しておきたいケースが出てくる。
そういった場合、以下のようにargparseのときのように@click.option()デコレータ内で指定することが可能。
type=<type>で指定。
@click.option('-m', '--minimum-length', 'minimum_length', type=int, default=18, help='Discard trimmed reads that are shorter than LENGTH. Default: 18')
パラメータタイプは、int、float、bool、strが指定できるようです。
http://click.pocoo.org/6/parameters/
デフォルトの値を決めておきたい
特定のオプションでデフォルト値を指定しておきたいケースがある。 その場合、デフォルト値の設定は以下のように行う。
default='hogehoge'で指定。
@click.option('-nl', '--nucleotide-trimming-list', 'nucleotide_trimming_list', default='A,N', help='Trimmed nucleotide list. Default: A,N')
指定する引数を限定したい
オプションの引数として、特定の文字列や数値しか受け付けたくない場合、以下のように指定できる引数を限定することができる。
type=click.Choice(['hoge', 'huga'])で指定。
@click.option('-nd', '--nucleotide-trimming-direction', 'nucleotide_trimming_direction', type=click.Choice(['three', 'five']), help='Trimmed nucleotide direction. Default: three')
Helpパラメータをカスタマイズしたい
Clickのデフォルトでは、Help文は--helpを指定することで出力される。
一般的に、Help文は--helpだけでなく-hでも出力されるケースが多い。そこで、-hでもHelp文が出力されるように設定を変更する。
@click.command()デコレータ内で指定。
CONTEXT_SETTINGS = dict(help_option_names=['-h', '--help']) @click.command(context_settings=CONTEXT_SETTINGS)
サブコマンド(後述)を利用している場合は、@click.groupデコレータ内で指定する。
CONTEXT_SETTINGS = dict(help_option_names=['-h', '--help']) @click.group(context_settings=CONTEXT_SETTINGS)
コールバック関数で値を検知・Helpを表示
オプションの引数を評価したい場合、コールバック関数を利用すると便利です。
@click.option()デコレータ内でcallback=<function名>を指定する。
また、コールバック関数内で、ctx.get_help()でHelp文を表示させることができる。ctx.exit()でPythonスクリプトの実行を止めることができる。
def validate_ifastq(ctx, param, value):
if not value:
click.echo('Error: Do not choose your FASTQ file.\n')
print(ctx.get_help())
ctx.exit()
return(value)
@cmd.command()
@click.option('-i', '--ifastq', 'ifastq', callback=validate_ifastq, help='Input file in FASTQ file. [required]')
サブコマンドを用意したい場合
@click.group()デコレータを用いて、サブコマンドを利用することができます。
#/usr/bin/env python
import click
@click.group()
def cmd():
pass
@cmd.command()
@click.option('-i', '--ifastq', 'ifastq', help='Input file in FASTQ file.')
@click.option('-o', '--output-prefix', 'output', help='Prefix of output file.')
def trim_polya(ifastq, output):
click.echo('Start trimming polyA sequence from reads...')
@cmd.command()
@click.option('-i', '--ifastq', 'ifastq', help='Input file in FASTQ file.')
@click.option('-o', '--output-prefix', 'output', help='Prefix of output file.')
def get_pass_read(ifastq, output):
click.echo('Start getting polyA site-supporting reads...')
def main():
cmd()
if __name__ == '__main__':
main()
Help文は以下のとおり。
$ python test.py --help Usage: test.py [OPTIONS] COMMAND [ARGS]... Options: --help Show this message and exit. Commands: get_pass_read trim_polya
サブコマンドごとのオプションをHelp文で出力することもできる。
$ python test.py get_pass_read --help Usage: test.py get_pass_read [OPTIONS] Options: -i, --ifastq TEXT Input file in FASTQ file. -o, --output-prefix TEXT Prefix of output file. --help Show this message and exit.
ENCODEプロジェクトのNGSデータを活用する
概要
ENCODEプロジェクトとは、ポストゲノム研究戦略としてヒトゲノムの機能性エレメントの同定とその全体像を解明するために組織された国際プロジェクトの1つです。
RNA-seq、ChIP-seq、DNase-seq、Hi-C、ChIA-PET、eCLIP-seqなどの解析手法を用いて、毎月膨大なデータを産出しています。
ENCODEプロジェクトでは、共通のプロトコルに基づいた実験と解析によりデータが産出され、即座に一般公開される仕組みを取っています。また、プロトコルも公開されているので自身で内容を確認・再解析することもできます。
ENCODEプロジェクトのデータは、プロジェクトに参加していないラボ・研究機関であっても、ウェブサイト上で公開されているNGSデータに関しては、自由にダウンロードして解析し論文のデータとして使用することもできます。
Data Use, Software, and Analysis Release Policies – ENCODE
実際に、ENCODEのデータを利用して投稿された論文はこれまで1700本にのぼります。
Publications using ENCODE data – ENCODE
ENCODEプロジェクトのデータベースには、有用な情報やデータがいっぱい公開されているのでこれらを使わない手はありません。
そこで今回は、ENCODEプロジェクトに登録されているデータを活用して手軽にデータ解析する方法をまとめました。今回は主に、RNA結合タンパク質(RBP)が結合するRNA領域を網羅的に同定する方法であるeCLIP-seqデータについて見ていきたいと思います。
また、RBPをIPした時に使用した抗体の情報など即座に使える情報も載っているのでそれらについても紹介しようと思います。
欲しいデータを探す
キーワードで検索
まず、ENCODEプロジェクトのウェブサイトに行きます。
ENCODE: Encyclopedia of DNA Elements – ENCODE
右上の検索欄に興味のあるRBPの名前を入力します。

試しにSFPQというRBPで検索にかけると、eCLIP-seq、Bind-n-seq、RNA-seq等のデータや、IPに使用した抗体の情報が出てきます。

データの種類から検索
他にも、[Encyclopedia]->[about]から各種データにアクセスできます。

この他にも、いろいろな検索方法が用意されていますが割愛します。
抗体の情報
右端にAntibodyと記載されているのが、抗体の情報です。
その中でも、緑色のマークがついているものは、ENCODEでStandardな抗体として認められた抗体です(抗体の特異性など幾つかの基準をもとに選定されている)。

クリックすると、その抗体でIPしたときのWestern blottingのデータや、shRNAでノックダウンしたときに目的のバンドが減弱しているかどうかWestern blottingで確認したデータなどが公開されています。

以上の情報から、各メーカーの抗体の良し悪しを知ることができます。 ちなみに、以下がENCODEプロジェクトで確認した抗体に関する論文になります。
http://yeolab.github.io/papers/2016/sundararaman_molcell_2016.pdf
Bind-n-seqデータ
Bind-n-seqについては、下記の論文を参照のこと。方法の詳細については、ここでは割愛します。
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4142047/
リコンビナントタンパク質のQCデータと、Enrichされたモチーフ配列の情報が載っています。

さらに、Processed dataの項目のところに、Enrichされた4, 5, 6, 7ntのモチーフ配列のデータがそれぞれ置いてあります(output typeがenrichmentとなっているやつ)。
ダウンロードボタン(Accessionにある小さいアイコン)をクリックすると、該当のファイルをダウンロードできます。
同じページにRawデータ(FASTQファイル)とプロトコルが置いてあるので、自身の手で再解析することも可能です。
eCLIP-seqデータ
1. 必要なファイルをダウンロード
eCLIPのデータを選択し、該当するデータのページに飛ぶと下のほうにProcessed dataの項目があります。
ゲノム上にマッピングされたリードを可視化するためのデータとしてbigWigファイルを、各Peakの染色体座標軸に関するデータとしてbedファイルをそれぞれダウンロードします。
また、右上のGRCh38(hg38)をhg19に変更することで、異なるバージョンのヒトのリファレンスゲノムをもとにして作成したファイルにアクセスできます。

今回は、hg38 (GRCh38)のデータをダウンロードします。
$ wget https://www.encodeproject.org/files/ENCFF823MPG/@@download/ENCFF823MPG.bigWig $ wget https://www.encodeproject.org/files/ENCFF960OTE/@@download/ENCFF960OTE.bed.gz $ gunzip ENCFF960OTE.bed.gz
ファイル名がわかりにくいのでリネームします。
$ mv ENCFF823MPG.bigWig SFPQ_eCLIP_rep1_ENCFF823MPG.bigWig $ mv ENCFF960OTE.bed SFPQ_eCLIP_rep1_ENCFF960OTE.bed
2. ファイルフォーマットの変換
bedgraphファイルの用意
bigwigファイルではUCSC genome browserにアップロードできないので、bedgraphファイルと呼ばれる形式にフォーマット変換します。
変換作業を行うために、今回はbigWigToBedGraphというプログラムを使います。
下記のURLから、目的のプログラムのバイナリファイルを入手します。
http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/
$ wget http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/bigWigToBedGraph $ chmod 755 bigWigToBedGraph
ダウンロードした後、実行権限を与え、パスの通っているディレクトリに入れます。
続いて、bigWigファイルをbedgraphファイルにフォーマット変換します。
# bigWigToBedGraph <bigWigファイル> <bedGraphファイル> $ bigWigToBedGraph SFPQ_eCLIP_rep1_ENCFF823MPG.bigWig SFPQ_eCLIP_rep1_ENCFF823MPG.bg
- 1つ目の引数: フォーマット変換したい
bigwigファイルを指定。 - 2つ目の引数: 出力される
bedGraphファイルの名前を指定。
次に、UCSC genome brower用にデータを整えます。ENCODEのデータの中にはハプロタイプのシークエンスにもマッピングしたデータが含まれているので、UCSC genome browserでデータを可視化するために、Chr1-22, chrX, chrY, chrMからなるCanonical genomeにマッピングされたデータのみを抽出する必要があります。
下記のような適当なスクリプトを書いて、データを整えます。
$ python ENCODE_eCLIP_mod.py SFPQ_eCLIP_rep1_ENCFF823MPG.bg SFPQ_eCLIP_rep1_ENCFF823MPG_for_UCSC.bg
最後に、UCSC genome browserへデータをアップロードする際に、ファイルの種類やTrackの名前などを提示する必要があります。それらの情報をヘッダー行(ファイルの一行目)に記載します。
$ echo "track type=bedGraph name=SFPQ_eCLIP_rep1_ENCFF823MPG description=SFPQ_eCLIP_rep1_ENCFF823MPG visibility=2 maxHeightPixels=40:40:20 color=255,0,0" > ./header_tmp.txt $ cat header_tmp.txt SFPQ_eCLIP_rep1_ENCFF823MPG_for_UCSC.bg > SFPQ_eCLIP_rep1_ENCFF823MPG_for_UCSC_header_plus.bg $ bzip2 -c SFPQ_eCLIP_rep1_ENCFF823MPG_for_UCSC_header_plus.bg > SFPQ_eCLIP_rep1_ENCFF823MPG_for_UCSC_header_plus.bg.bz2
echoコマンドのところでヘッダー行の情報をtxtファイルとして出力しています。
そのあとに、catコマンドを用いて、先ほど作ったBedGraphファイルと結合させます。
出力されたファイルをそのままアップロードしてもOKですが、容量が大きいためアップロードに時間がかかります。そのため、アップロードするファイルのサイズを小さくするために、ここではbzip2ファイルに圧縮しています。
bedファイルの用意
ENCODEのサイトから得られるbedファイルを、bigWigファイルと同様にUCSC genome browserにアップロードできるファイルに加工します。
下記のような適当なスクリプトを書いて、データを整えます。
$ python ENCODE_eCLIP_mod_bed.py SFPQ_eCLIP_rep1_ENCFF960OTE.bed SFPQ_eCLIP_rep1_ENCFF960OTE_for_UCSC.bed
最後に、UCSC genome browserへデータをアップロードするために、ファイルの種類やTrackの名前の情報をヘッダー行(ファイルの一行目)に記載します。ここも先程と同様です。
$ echo "track type=bed name=SFPQ_eCLIP_rep1_ENCFF960OTE_peak description=SFPQ_eCLIP_rep1_ENCFF960OTE_peak visibility=2 maxHeightPixels=40:40:20 color=255,0,0" > ./header_tmp.txt $ cat header_tmp.txt SFPQ_eCLIP_rep1_ENCFF960OTE_for_UCSC.bed > SFPQ_eCLIP_rep1_ENCFF960OTE_for_UCSC_header_plus.bed $ bzip2 -c SFPQ_eCLIP_rep1_ENCFF960OTE_for_UCSC_header_plus.bed > SFPQ_eCLIP_rep1_ENCFF960OTE_for_UCSC_header_plus.bed.bz2
データのアップロード
UCSC genome browserにデータをアップロードします。
ホーム画面から、[MyData] -> [Custom Tracks]をクリックします。
Add Custom Tracksというページに移動するので、ファイルを選択をクリックし、アップロードするファイルを選択し、submitボタンをクリックします。
アップロードが完了すると、Manage Custom Tracksというページに移動します。右端にあるgoボタンをクリックすると、データをみることができます。

可視化したデータをみてみると、以下のようになります。

ざっくりとですが、以上になります。
HoloLensを使ってPDBに登録されているタンパク質の立体構造を現実空間に投影する
概要
HoloLens向けのアプリをビルドして実機にデプロイするところをまとめてみました。
Microsoftが用意しているHello World的なチュートリアルをなぞってみます。
Holograms 100
ただのCubeを表示させるだけだと味気ないので、
PDBからタンパク質の立体構造のデータを取ってきて、PyMolとBlenderで適当に加工した3Dデータを表示させます。
動画をキャプチャすると見た目がしょぼく見えてしまいますがこんな感じになります。
このように、HoloLensを使えば、空間を認識してマーカーなしでその場にオブジェクトを置くことができます。
必要なもの
HoloLensのアプリ開発環境
- Visual Studio 2015 Update 3
http://dev.windows.com/downloads - HoloLens Emulator
http://go.microsoft.com/fwlink/?LinkID=823018 - Unity 5.5
https://store.unity.com/download
PDBデータの加工
- Anaconda3 (Python3.5, Scipy)
- PyMOL
- Blender 2.78a
https://www.blender.org/download/
内容
1. HoloLensのアプリ開発に必要なツールのインストール
詳しくは、MicrosoftのInstall the toolsを参照のこと。
https://developer.microsoft.com/ja-jp/windows/holographic/install_the_tools
- Visual Studio 2015 Update 3
http://dev.windows.com/downloads - HoloLens Emulator
http://go.microsoft.com/fwlink/?LinkID=823018 - Unity 5.5
https://store.unity.com/download
をそれぞれインストールする。
インストール時の留意点
Visual Studio 2015 Update 3
Tools (1.4)とWindows 10 SDK (10.0.10586)を併せてインストールする必要がある。
HoloLens Emulator
Hyper-Vの機能をあらかじめONにしておく必要がある。
方法は以下の通り。
1. CortanaでHyper-Vと検索。
2. Windowsの機能の無効化と有効化(コントロールパネル)をクリック。
3. Hyper-Vの項目にチェックを付け、パソコンを再起動

Unity
Windows Store .NET Scripting BackEndを併せてインストールする。
2. PDBデータの加工に必要なツールのインストール
Anacondaのインストール
PyMolがPythonで動作するソフトであるため、Python3をあらかじめパソコンにインストールしておく。
Download Anaconda Now! | Continuum
Python2.7でも構わないが、今回はPython3.5のほうをダウンロードしてインストールした。
PyMOLのインストール
下記の記事に紹介されているので詳しくはそちらを参照のこと。
PyMOL インストール
1. 非公式のPythonパッケージのインストーラーを配布しているサイトへ行き、
Python Extension Packages for Windows - Christoph Gohlke
- Pmw-2.0.1-py3-none-any.whl
- pymol-1.8.5.0-cp35-cp35m-win_amd64.whl
- pymol_launcher-1.0-cp35-cp35m-win_amd64.whl
をそれぞれダウンロードする。

2. コマンドプロンプトを開き、先ほどダウンロードしたファイルからPyMOLをインストールする。
$ pip install Pmw-2.0.1-py3-none-any.whl $ pip install pymol-1.8.5.0-cp35-cp35m-win_amd64.whl $ pip install pymol_launcher-1.0-cp35-cp35m-win_amd64.whl
Anacondaをインストールしていれば、ScipyとWheelがインストールされているはずですが、万が一上記のライブラリがインストールされていない場合は下記のように自身でインストールする。

Anaconda Navigatorを起動して、Environmentsをクリックし、Allを選択して、それぞれのライブラリを検索する。 インストールしたいライブラリにチェックを付け、右下のApplyボタンをクリックして該当するライブラリをインストールする。
3. PyMOLの実行ファイルのショートカットを作っておく。
PyMOLはC:\Users\自分のユーザー名\Anaconda3\PyMOL.exeにあるので、ショートカットを作っておいてデスクトップなどに置いておくと便利です。

Blenderのインストール
下記のURLからインストーラーをダウンロードし、自身の環境にインストールする。
https://www.blender.org/download/
3. Unityに取り込ませるタンパク質の立体構造のデータを用意する
PDBのサイトから適当なタンパク質の立体構造をダウンロード
下記のURLからPDBのサイトへアクセスします。
RCSB Protein Data Bank - RCSB PDB
PDBのサイトから、好きなタンパク質の名前で検索をかけ、適当なタンパク質のデータを選択する。
各立体構造のページに飛んだら、右上のDownload Filesボタンをクリックして、PDB Formatのファイルをダウンロードする。

ちなみ、今回使用したデータはこちら。
https://files.rcsb.org/download/3Q0L.pdb
PyMOL上でリボン構造とSurface構造を保存する
PyMOLをWindows10上で起動し、[File]->[Open]から先ほどダウンロードしてきたPDBファイルを開く。

開いたデータ(ここでは「3qOl」)の欄の横にあるHボタンをクリックし、メニューの先頭のeverythingを選択する。

すると、右図のように立体構造の表示がクリアされる。
次に、Sボタンをクリックし、メニューのcartoonを選択する。

すると、リボン構造としてタンパク質の立体構造が表現される。
これをBlenderで取り込めるVRML2ファイルとして保存する。

Surface構造も同様の手順で取得する。
まず、先ほどと同様にHボタンをクリックし、メニューの先頭のeverythingをクリックすることでリボン構造を消す。
次に、Sボタンをクリックし、メニューの先頭のsurfaceを選択する。

すると今度は、Surface構造としてタンパク質の立体構造が表示される。
これをBlenderで取り込めるVRML2ファイルとして保存する。
Blenderで3Dデータを加工する
Blenderを起動する。起動した初期画面にはCubeがあらかじめ設置されているので、まずはこれを消す。 右クリックでオブジェクトを選択して、キーボードの「Delete」ボタンを押すと、ウインドウ上に「Delete」するか求められるので「Delete」をクリックしてCubeを消す。

BlenderがVRML2ファイルを取り込めるかどうか確認する。
[File]->[User Preferences]をクリックして、Blender User Preferencesを開く。
Add-onsのタブをクリックし、VAML2を検索する。Web3D X3D/VAML2 formatの項目にチェックが付いていない場合は、チェックを付けて左下のSave User Settingsをクリックする。

次に、Blenderにタンパク質の立体構造のデータ(リボン構造とSurface構造)を取り込ませる。
[File]->[Import]->[X3D Extensible 3D(.x3d/.wrl)]をクリックする。そして、先ほどPyMOLで用意したリボン構造のほうのVAML2ファイルを選択しBlenderにインポートする。

- 取り込んだリボン構造に対して右クリックし、オブジェクトを選択する。
- キーボードの
Tabキーを押して、Object ModeからEdit Modeに切り替える。 - 右端のカラムから
Materialボタンを押す。 +ボタンをクリックして、新しいMaterialMaterial.001を作成する。- 新しく作ったMaterialを選択して、
Assignボタンをクリックする。 - Diffuseの項目で適当な色を選択する。
Tabキーを押して、Object Modeに戻る。- もとからあった
Shapeを選択し、-ボタンをクリックして消す。

結果として、このようにリボン構造に色がつくはずです。
同じ要領でSurface構造をBlenderにインポートする。
- 先程と同様に、右クリックでSurface構造のみを選択した状態で、
Tabキーを押しEdit Modeに切り替える。 - 右端のカラムから
Materialボタンを押す。 - Diffuseの項目で適当な色を選択する。
Transparencyにチェックを付ける(リボン構造と異なり、Surface構造は透明になるように設定します)。Alpha値を0.3に設定する(透明度を設定。0に近いほど透明度が増す)。Tabキーを押し、Object Modeに切り替える。- キーボードの
Aキーを押し、オブジェクトを全選択。 Sキーを押し、マウスを動かしてオブジェクトのサイズが小さくなるように調節する。 もとのサイズだと保存したファイルが大きくなるので、できるだけ容量を抑えるためにサイズを小さくします。

最後に、完成したオブジェクトを、UnityにインポートできるFBXファイルとしてエクスポートする。
[File]->[Export]->[FBX(.fbx)]をクリックし、任意の場所・名前で保存。

4. Unity上でHoloLensアプリをつくる
新しいプロジェクトの作成
Unityを起動して、右上のNEWをクリック。プロジェクト名をPDBViewerとして、Create projectボタンをクリックして、新しいプロジェクトを作成する。

タンパク質の立体構造のデータをインポート
上記で作成したタンパク質の立体構造のFBXファイルをUnityにインポートする。
方法は簡単で、ProjectタブのAssetsの直下に、該当のFBXファイルをドラッグ&ドロップでインポートする。

[Ctrl]+[S]で新しいSceneを適当な名前で保存する。今回はPDBViewerという名前で保存した。
HoloLens向けにMain cameraの設定
次に、HoloLens向けにMain cameraの設定(Inspector)を変更する。
- TransformのPositionを
(X: 0, Y: 1, Z: -10)から(X: 0, Y: 0, Z: 0)に変更。 - CamemraのClear Flagsを
SkyboxからSolid colorに変更。 - CameraのBackgroundのRGBA値を
(0, 0, 0, 0)に変更。 - CameraのNear Clip Planeを
0.3から0.85に変更(85 cmより近い地点ではオブジェクトの表示を消す)。

タンパク質の立体構造のデータをSceneに追加
Assetsから、Hierarchyにタンパク質の立体構造のデータをドラッグ&ドロップで配置する。

TranformでPositionを(X: 0, Y: 0, Z: 0.5)、Scaleを(X: 0.1, Y: 0.1, Z: 0.1)に設定する。

HoloLens向けにビルドするための設定変更
1. Unity Performance settings
[Edit]->[Project Settings]->[Quality]をクリックして、Qualityの設定画面を出す。

Defaultの横にある下三角ボタンをクリックし、Fastestを選択する。

QualityをFantasticからFastestにしておかないとFPSの低下を招き、結果としてHoloLens上での動作がもたつく。
2. Unity Build settings
HoloLens向けのビルドの設定を行う。
まず、[File]->[Build Setting…]をクリックして、ビルドの設定画面を出す。

- SDKを
Universal 10に変更。 - UWP Build Typeを
D3Dに変更。 Unity C# Projectsにチェックを付ける。
最後に、Windows Storeを選択して、左下のSwitch Platformをクリックする。
すると、UnityのマークがWindows Storeに移動し、Build対象が切り替わったことが確認できます。

Player Settings...をクリックし、Settings for Windows StoreのOther settingsのVirtual Reality Supportedにチェックを付ける。
Virtual Reality SDKsの項目で、Windows Holographicを選択する。

先ほど行ったビルドの設定を先にしておかないと、Virtual Reality Supportedの設定でWindows Holographicが選択肢に出てこないので注意。
3. ビルドを行う
[File]->[Build Settings…]をクリックし、ビルドの設定画面を開き、Buildボタンをクリック。
どこにアプリをビルドするか聞かれるので、Appフォルダを新規作成してそこにアプリをビルドする。

5. Visual StudioからHoloLensへアプリをデプロイする
ビルドが終わるとプロジェクトのウインドウが開くので、AppフォルダからPDBViewer.slnをクリックする。

Visual Studioが起動する。Package.appxmanifestを右クリックして、View Codeをクリックする。

TargetDeviceFamilyの名前のところをWindows.UniversalからWindows.Holographicに変更し、ファイルを保存。

HoloLensをUSB経由でパソコンに接続し、Visual Studio上で以下のように設定する。
DebugをReleaseに変更。ARMをx86に変更。Local MachineをDeviceに変更。
[Debug]->[Start Without Debugging]をクリックし、HoloLensにアプリをデプロイする。

以上になります。よくよく見ると今回プログラミングを一切していません…。
参考
詳しくはこちらを参照のこと。
Windows10でNGS解析をやってみる - Bash on Ubuntu on Windows × Bioconda
概要
次世代シーケンス(NGS)のデータ解析というとLinux OS上で作業するのが一般的で、Windowsで行うためにはVMwareやVirtualBoxでLinuxの仮想環境を構築する必要がありました。
ただ、この方法だと環境構築が面倒で、場合によってはうまく動かないケースがありました。また、LinuxとWindowsの両方のシステムを動かすことになるので、パソコンへの負荷が大きいのも欠点でした。
しかし、2016年8月3日に配信開始された大型アップデート「Windows 10 Anniversary Update」で状況が変わりました。
このアップデートでLinux(Ubuntu)がWindows上で使用可能になる「Windows Subsystem for Linux (WSL)」が追加され、「Bash on Ubuntu on Windows」と呼ばれるBashがWindows上でも使えるようになりました。
今回はこのBash on Ubuntu on WindowsとBiocondaを組み合わせることで、NGSのデータ解析を行ってみたいと思います。
内容
1. Bash on Ubuntu on Windowsのインストール
下記のブログ記事ですでに紹介されているのでそちらを参照のこと。
インストール後、以下のようにメニュー画面からBash on Ubuntu on Windowsを呼び出すことができるようになります。

起動するとこんな感じです。

2. Biocondaのインストール
以前のブログで記事ですでに紹介したのでそちらを参照のこと。
Bash on Ubuntu on Windows上でもminicondaやBiocondaをインストールすることができます。

Bash on Ubuntu on WindowsからWindows内のフォルダにアクセスする
Bash on Ubuntu on Windows上からだと、WindowsのCドライブなどが/mnt上にマウントされているように見えます。

$ cd /mnt/c $ cd /mnt/d
などとそれぞれのドライブにアクセスできます。
もちろん、Windowsのシステムファイルに直接アクセスすることはできませんが、ファイル操作などは行うことができます。 そのため、LinuxのサブシステムとWindows10の間でのファイル共有も容易です。
$ touch test.txt $ mv test.txt /mnt/d

RNA-seqのデータをいじってみる
Biocondaを使って、sra-toolsとfastqcをインストールします。
$ conda install sra-tools $ conda install fastqc
今回、下記のRNA-seqのSRAファイルを使用する。
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSM1606099
Bashのホームディレクトリではなく、Dドライブ上にデータを配置するため、Dドライブに移動する。
cd /mnt/d
Bash on Ubuntu on Windows上でSRAファイルをダウンロードする。
$ wget ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByExp/sra/SRX/SRX870/SRX870796/SRR1795409/SRR1795409.sra
SRAファイルを解凍してFASTQファイルを用意する。
fastq-dump SRR1795409.sra
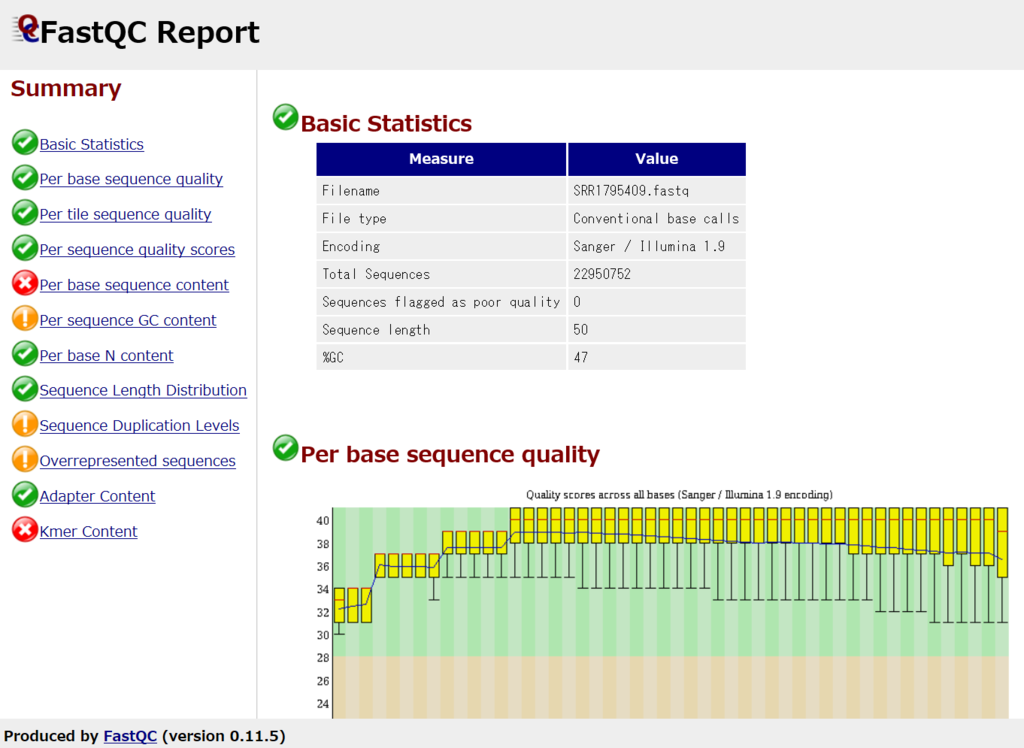
FastQCを使って、FASTQファイルの中身をチェックする。
fastqc SRR1795409.fastq
Dドライブ上にFastQCのデータが保存されているので、Windows上からアクセスしてファイルの中身を見ることができます。

こんな感じ。
ガッツリ解析しようと思ったらスパコンを利用するほうが良いと思いますが、Windows上にテスト環境を作って練習するには手軽かもしれません。
NCBI GEOからまとめてSRAファイルを取得する
概要
NCBI GEOからまとめてSRAファイルを取得したい。
準備
Entrez Directのコンパイル済みのバイナリファイルをダウンロードして、パスを通すだけ。
* Entrez Direct
ftp://ftp.ncbi.nlm.nih.gov/entrez/entrezdirect/
https://www.ncbi.nlm.nih.gov/news/02-06-2014-entrez-direct-released/
インストール例
$ wget ftp://ftp.ncbi.nlm.nih.gov/entrez/entrezdirect/edirect.tar.gz
$ tar zxvf edirect.tar.gz
$ echo 'export PATH=~/softwareedirect:${PATH}' >> ~/.bashrc
もしくは、biocondaでインストールする。
$ conda install entrez-direct
実行例

下記のコマンドを実行。
esearchの-queryオプションに適当なBioProjectのIDを指定する。
$ esearch -db sra -query PRJNA328218 | efetch --format runinfo | cut -d ',' -f 10 > srafile.txt $ wget -i srafile.txt
SRAファイルのダウンロード先を取得して、wgetでまとめてダウンロードする。
esearchの-dbオプションからさまざまなデータベース(SRA、PubMedなど)を指定でき、-queryオプションでIDなどをもとに検索を行うことができる。
efetchで検索した項目の情報を取得することができる。--formatの指定は、runinfoとnativeの2種類ありruninfoではカンマ区切りで各サンプルの情報が出力される(すべての情報が得られるわけではなく概要のみ?)。
一方、nativeを指定すると、XMLファイル形式ですべての情報が出力される(すべての情報が揃っているが、中身を調べるにはXMLをパースして整理する必要がある)。
SRAのファイル名にサンプル名を加えたい
SRAファイルをダウンロードした後、SRA IDとサンプル名を併記したファイル名にリネームする。
シェルスクリプト
以下のようにスクリプトを実行する。
./sra_download.sh <任意のBioProjectのID>
実行に必要なPythonスクリプト
parse_sra_xml.py (1.4 kB)
rename_sra_files.py (352 B)
使用する場合、
filepath="/path/to/python_scripts"
の部分に実行に必要なPythonスクリプトを置いた場所を指定する。
やっていることは単純で、
1. 特定のProject IDに含まれる複数のサンプル(SRAファイル)をダウンロード&XMLファイルを取得する。
2. XMLファイルをパースして、SRAファイル名とサンプル名の対応表を作る。
3. SRA IDとサンプル名が併記されたファイル名にリネームする。
という作業をPythonのスクリプトも使いながら(スマートじゃないですが)行っています。
実行すると、以下のようにSRAファイルが一括ダウンロードできます。

参考
- Tutorial: How to download raw sequence data from GEO/SRA
https://www.biostars.org/p/111040/ - NCBI SRA から FASTQ をダウンロードする方法
http://bi.biopapyrus.net/transcriptome/rnaseq-data/download-from-sra.html - prefetch すらっと落とす SRA
http://blog.amelieff.jp/?eid=231191
biocondaを利用してNGS関連のソフトウェアを一括でインストールする
概要
biocondaと呼ばれるパッケージマネージャーを用いて、バイオインフォマティクス関連のソフトウェアのインストールから、バージョン管理までを行うことができます。
バイオインフォマティクスの解析環境を用意しようと思っているなら、自力でソフトウェアをインストールするのではなく、biocondaを利用したほうが圧倒的に楽です(MEMEやCircosなどインストールに手間取るソフトウェアが一行のコマンドでインストールできちゃう)。
biocondaには現在、15,000以上のBiology関連のソフトウェア・パッケージが登録されており、おおむね、どのソフトウェアに関しても最新バージョンがインストールできる環境が整備されているみたいです。
ただし、パッケージが充実しているのはLinux版で、MacOS版はbiocondaの(インストールの)レシピが充実していない印象です。 なので、biocondaを導入する対象はLinux OSであることが望ましいと思います。

内容
基本的に、こちらのbioconda公式サイトにインストール方法が記述されているので、詳しくはこちらを参照のこと。
https://bioconda.github.io/
インストール方法
1. minicondaをインストールする(Anacondaというのもありますが、minicondaのほうはその最小構成版になっています。余計なパッケージをはじめから入れさせないためにminicondaを選択したほうがよいかもしれません)。
Python3がおすすめと書いてあるが、NGS関連のソフトウェアはpython2でコーディングされているものも多いため、python2の方を選択する。
http://conda.pydata.org/miniconda.html

2. 上記のURLからシェルスクリプトをダウンロードし、実行する。
ここでは、64bit版のPython2の環境を選択してインストールしています。
$ wget https://repo.continuum.io/miniconda/Miniconda2-latest-Linux-x86_64.sh $ bash Miniconda2-latest-Linux-x86_64.sh
いろいろ聞かれるが、基本的にEnterを押すか、Yesを入力すればいい。
最終的に、カレントディレクトリにminiconda2というディレクトリができており、.bashrcに以下の文が追加されているはずです。
> .bashrc # added by Miniconda2 4.2.12 installer export PATH="/home/user/miniconda2/bin:$PATH"
最後の質問で、Noを選択すると、上記の文が追加されません。
その場合、自身で上記の文を記述して、miniconda2/binにパスを通す必要があります。
3. インストール終了後、.bashrcを再読み込み。
$ source ~/.bashrc
4. condaが正常にインストールされたかどうかチェック。 エラーが返ってこなければOK。
$ conda -h
5. biocondaのチャンネルを追加。biocondaチャンネルが最上位に来るように設定。
$ conda config --add channels conda-forge $ conda config --add channels defaults $ conda config --add channels r $ conda config --add channels bioconda
6.任意のパッケージ・ソフトウェアをインストール。
$ conda install cutadapt $ conda install meme $ conda install fastqc $ conda install fastx_toolkit $ conda install bowtie $ conda install bowtie2 $ conda install prinseq $ conda install circos $ conda install tophat $ conda install cufflinks $ conda install samtools $ conda install bedtools $ conda install sra-tools $ conda install trimmomatic $ conda install paralyzer $ conda install star $ conda install rsem $ conda install bwa $ conda install picard $ conda install subread $ conda install entrez-direct $ conda install art $ conda install macs2 $ conda install htseq $ conda install rseqc
biocondaに登録されているパッケージ群のリストは下記のサイトからチェックできる。
https://bioconda.github.io/recipes.html
インストール時の注意点
biocondaをインストールする際に、PYTHONPATHが.bashrcに記載されていると、うまくインストールされない。
インストールチェックを行うと、エラーが発生する。
$ conda -h
Traceback (most recent call last):
File "/home/akimitsu/miniconda3/bin/conda", line 4, in <module>
import conda.cli
ImportError: No module named 'conda'
対処法としては、.bashrc中でPYTHONPATHをExportしている箇所をコメントアウトする。ターミナルを再起動・再ログインしてから、再インストールする。
# コメントアウト
# export PYTHONHOME=/usr/local/package/python2.7/current
# export PYTHONPATH=/home/user/software/python_path27/lib/python2.7/site-packages
# export PATH=/home/user/software/python_path27/bin:${PYTHONHOME}/bin:${PATH}
# export LD_LIBRARY_PATH=/home/user/software/python_path27/lib:${PYTHONHOME}/lib:${LD_LIBRARY_PATH}
biocondaに登録されているパッケージのインストールに関する注意点
python2に依存しているパッケージは、python3環境下ではインストールできない。 そのため、以下で説明するPython2用の仮想環境を構築するか、python2向けのminicondaを再インストールする必要がある。
$ conda install macs2 Fetching package metadata ............. Solving package specifications: .... UnsatisfiableError: The following specifications were found to be in conflict: - macs2 - python 3.5* Use "conda info <package>" to see the dependencies for each package.
python2に依存しているパッケージ例
conda install macs2 conda install htseq conda install rseqc
biocondaの環境をチェック
biocondaでインストールしたパッケージは、conda listで確認できる。
$ conda list # packages in environment at /home/akimitsu/miniconda2: # art 3.19.15 1 bioconda bcftools 1.3.1 1 bioconda bedtools 2.26.0 0 bioconda bowtie 1.2.0 py27_0 bioconda bowtie2 2.3.0 py27_0 bioconda bwa 0.7.15 0 bioconda bx-python 0.7.3 py27_0 bioconda bzip2 1.0.6 3 cairo 1.14.6 0 cffi 1.9.1 py27_0 circos 0.69.2 0 bioconda ...
python2向けのbiocondaでpython3を使う
python3を動かすための仮想環境を構築することができる。
以下では、py35という名前のpython3.5がインストールされた仮想環境を作っている。
$ conda create --name py35 python=3.5
--nameで仮想環境の名前を指定、
python=でPythonのバージョンを指定する。
condaでは現在、Python 2.7, 3.4, 3.5に対応している。
Managing Python — Conda documentation
登録した環境設定を確認する。
$ conda info --envs # conda environments: # py35 /home/your_name/miniconda2/envs/py35 root * /home/your_name/miniconda2
環境の切り替え。
# py35に入る source activate py35 # py35から抜ける source deactivate py35
これを利用して、プロジェクトごとに環境を用意して特定のパッケージ・バージョンで解析を行うこともできます。
CRANに登録されているRパッケージをインストールする
biocondaに登録されていないPythonやRのパッケージは、pipやinstall.packages()で正常にインストールできないことがある。 とりあえず、Rのパッケージは以下の通りに入力するとインストールされるみたいです。
$ R
> install.packages('任意のパッケージ', repos='http://cran.us.r-project.org')
参照
- bioconda
https://bioconda.github.io/
Leap Motionを使ってVR上にデスクトップ画面を作ろう
はじめに
Leap Motionとは
Leap Motionは、手の位置や指一本一本の動きをトラッキングすることのできる赤外線センサーです。
VR向けに対応したOrion SDKを導入することにより、Oculus RiftやHTC Viveと組み合わせてVR空間で手をトラッキングすることも可能になりました。
特徴
長所
- 手に握るタイプのコントローラー(Oculus touchなど)と違い、ものを持たずにVR上で操作が可能。
- ジェスチャーを使った直感的な操作ができる。
短所
- 手に握るタイプのコントローラーと比較して、トラッキング精度が劣る。
- ボタンのついたコントローラーと異なり、フォースフィードバックが感じられないので物を掴んだりする際に違和感を感じる。
- 視野角130度ぐらいしかカバーできないので、ジェスチャーでPinch outさせたときに手がトラッキングから外れることがしばしば。
- VRHMDを赤外線センサーでトラッキングするシステムを使っているHTC Viveだと、ViveとLeap Motionのセンサー同士が干渉することがある。
今回の目的
- HTC ViveとLeap Motionを用いて、VR空間にデスクトップ画面を生成する。
具体的には、親指と人差し指をくっつけた状態(Pinch point)で両手のPinch pointを近づけたときに、デスクトップ画面をVR空間内に生成させるというもの。
以下がそのイメージ図になります。

デモ
必要なもの
ハードウェア
- HTC Vive
- Leap Motion
使用したミドルウェア・アセットなど
- Unity5.5.0f3
- Leap Motion SDK v3.2.0 (ORION BETA)
- LeapMotion_CoreAsset_Orion_4.1.5.unitypackage
- Attachments-1.0.4.unitypackage
- UIInputModule-1.2.0.unitypackage
- uDesktopDuplication-v1.5.1.unitypackage
- SteamVR Plugin
- Purple Space Nebula Skybox
内容
Leap MotionのUnityアセットをあらかじめ、下記のサイトからダウンロードしておく。
Unity — Leap Motion Developer
デスクトップをミラーリングするのに、uDesktopDuplicationを使用した。 github.com
(1) SteamVR Pluginをインポートする。SteamVR_settingsが出てくるのでAccept Allをクリック(You made the right choice!というコメントが出る)。

(2) [Assets] -> [Import Package] -> [Custom Package...]をクリックして、
- LeapMotion_CoreAsset_Orion_4.1.5.unitypackage
- uDesktopDuplication-v1.4.2.unitypackage
をそれぞれインポートする。

(3) Scenesフォルダを作成し、新しいシーンPinchTestを用意する。
(4) HierarchyのMain cameraを削除する(VRHMD専用のCameraをあとで追加するので、Unityの既存のカメラを削除する必要がある。)
(5) Assets/LeapMotion/PrefabsからLMHeadMountedRig.prefabをHierarchyにドラッグ&ドロップする。

(6) Hierarchy中のLMHeadMountedRig/HandModelsにあるCapsuleHand_LとCapsuleHand_Rをそれぞれ右クリックし、Create emptyから空の子オブジェクトをつくる。それぞれ、PinchDetector_L、PinchDetector_Rと名前をつける。
(7) Assets/LeapMotion/Scripts/DetectionUtilitiesからPinchDetector.csを先ほど作成したPinchDetector_LとPinchDetector_Rにそれぞれアタッチする。これらのオブジェクトは人差し指と親指をくっつけた状態(Pinch)を手が取っているかどうか確認するのに必要です。

ここまででPinchの検出がする準備が整いました。
さらに、Pinch状態になっているかどうか視覚的に確認するために、ここではLeap Motionから提供されているAttachmentsのUnityアセットを導入します。
(8) [Assets] -> [Import Package] -> [Custom Package...]をクリックして、Attachments-1.0.4.unitypackageとUIInputModule-1.2.0.unitypackageをインポートする。
(9) Assets/LeapMotionModules/Attachments/PrefabsからHandAttachment_L.prefabとHandAttachment_R.prefabをHandModelsにアタッチする。

(10) 先ほどアタッチしたHandAttachmentsを、使用するHandModelsとして設定するためにLeapHandControllerの設定を変更する。
- Hand PoolのModel Poolで
Sizeを1から2に変更する。 - Model Poolが1つ追加されるので、
Group NameをAttachmentと記載し、Left ModelとRight ModelにHandAttachments-L/Rをそれぞれアタッチする。

(11) Assets/LeapMotionModules/UIInput/TexturesをCircleCursorをアタッチする。

(12) CircleCursorのTransformを書き換える。Positionを(0, 0, 0)、Rotationを(120, 0, 0)、Scaleを(0.15, 0.15, 0.15)に設定する。CircleCursorの位置を設定しているだけなので、適当な値を入れて構わない。

(13) PinchPointのInspectorから、Add Componentをクリックして、Pinch Detectorを選択する。

(14) Scriptsフォルダを作り、PinchCursor.csという名前のスクリプトを用意する。
Pinchを検出したときに、子オブジェクトをActive状態にするスクリプトになります。
(15) 先ほど作成したPinchCursor.csをPinchPointにアタッチする。

(16) PinchPointのInspectorのPinch Detector内にあるOn Activate()の+をクリック。
Pinch状態にあるとき、On Activate上で指定した関数が実行されます。一方で、Pinch状態が解除された場合、On Deactive上で指定した関数が実行されます。つまり、手の状態の遷移に伴って、さまざまな命令を実行ができるというわけです。
(17) PinchPointオブジェクトを図に示した箇所にドラッグ&ドロップ。
(18) No Functionをクリックし、先ほど作成したPinchCursorのpinchOn()関数を指定する。On Deactivate()に関しても同様の操作を行い、No FunctionにpinchOff()関数を指定する。

最終的に、以下のようになるはずです。

(19) CircleCursorオブジェクトのチェックを外して、Deactivateの状態にしておく。
こうしておくことで、シーンのはじめにCircleCursorが見えない状態になります。
逆にチェックを外しておかないとはじめからCursorが見えてしまいます。

(20) シーンのプレビューを行う。
以下のように、Pinchが検出されると、設定したCircleCursorが出現します。Pinch状態が解除されると、Cursorは見えなくなります。

最後に、Pinch状態にある両手を近づけたときに、デスクトップ画面がVR上で生成される仕組みを作っていきます。
(21) Hierarchy上で、何も選択していない状態で右クリックする。Create emptyを選択し、DisplayAnchorという名前の空のゲームオブジェクトをつくる。

(22) Scriptsフォルダ内にCreateDisplay.csという名前のスクリプトを作る。
(23) CreateDisplay.csスクリプトを先ほど作成したDisplayAnchorオブジェクトにアタッチする。

(24) Pinch Detector_L、Pinch Detector_R、Magic Display Prefabに、HandModels内のPinchDetector_L、PinchDetector_RとAssets/uDesktopDuplication/Examples/PrefabsのMonitor_Board.prefabをそれぞれアタッチする。

(25) Skyboxがデフォルトのままで物悲しい感じなので、UnityアセットのPurple Space Nebula Skyboxを導入する。

(26) Assets/Skyboxes/PurpleNebulaにあるPurpleNebula.matをシーンにドラッグ&ドロップ。

周りが宇宙空間になりました。少しはマシな感じになったと思います。

(27) 最後に、CenterEyeAnchorのInspectorのCameraでClear FlagsをSkyboxに変更する。
これを設定しないと、Skyboxが反映されないので周りが真っ暗なままになってしまいます。

(28) シーンのプレビューを行う。
以下のように両手のPinchを検出され、PinchPointを近づけたときにディスプレイが生成されます。
Pinch inもしくはPinch outすることで自由自在に大きさを変えられます。
手(親指と人差し指)を離すと、その場にディスプレイが固定されます。
以上、C#のスクリプトの記述量は少なめで、Unityアセットを多用してイメージ通りのものが作れました。
